Numerische Rechenmethoden etablieren sich mehr und mehr als ein drittes Standbein der Naturwissenschaften neben theoretischem und experimentellem Arbeiten. Moderne Computer sind in der Lage, außerordentlich komplexe Systeme aus so verschiedenen Bereichen wie Kosmologie, Evolution, Klimaforschung, oder Teilchenphysik mit anspruchsvollen Modellen zu beschreiben.
Für Chemiker und Lebenswissenschaftler sind insbesondere quantenmechanische Berechnungen zur Elektronenstruktur von Molekülen und Kraftfeldrechnungen zur Beschreibung der konformationellen Dynamik von Biomolekülen wichtig. Am Institut für physikalische Chemie arbeite ich als einer der Fortentwickler des Amber-Moleküldynamikpaketes http://www.ambermd.org/ . Dabei liegt der Schwerpunkt auf der Anwendung moderner Rechenmethoden in diesen beiden Gebieten:
Elektronenstruktur und Dynamik großer System
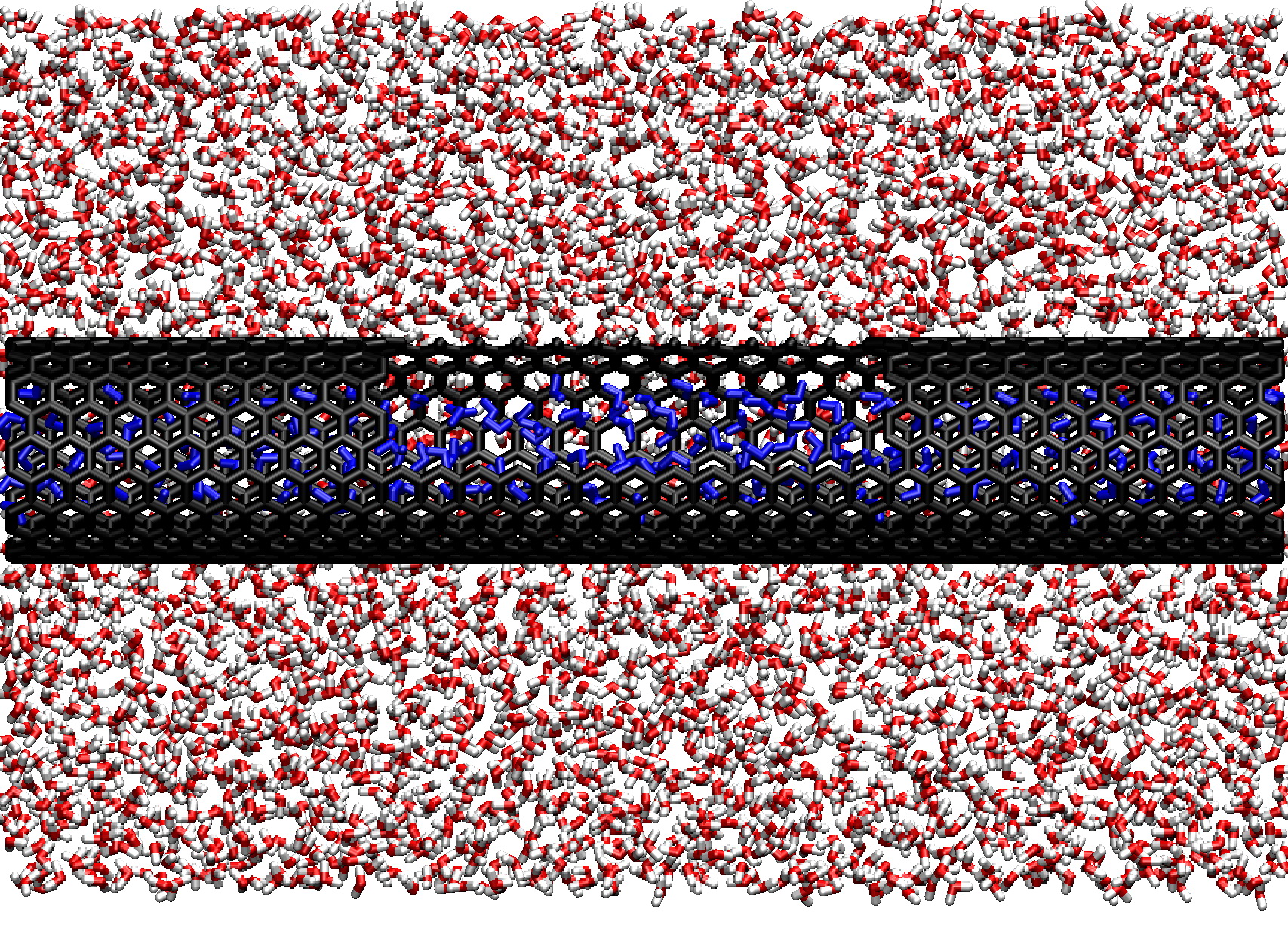 Die elektrischen Eigenschaften von Nanomaterialien sind von großem Interesse für die Entwicklung neuer Werkstoffe und miniaturisierter elektronischer Bauteile. Da diese Systeme experimentell sehr schwierig oder aufwändig experimentell zu untersuchen sind, können Computersimulationen hier zum besseren Verständnis ihrer Eigenschaften beitragen. Um die Elektronenstruktur von Molekülverbänden aus tausenden von Atomen wie Kohlenstoffnanoröhren zu beschreiben, kann man einfache quantenmechanische Methoden, gekoppelt mit Moleküldynamiksimulationen, anwenden. Damit lässt sich z.B. die Polarisation des Systems bei Ladungstransportphänomenen untersuchen und seine Leitfähigkeit berechnen.
Die elektrischen Eigenschaften von Nanomaterialien sind von großem Interesse für die Entwicklung neuer Werkstoffe und miniaturisierter elektronischer Bauteile. Da diese Systeme experimentell sehr schwierig oder aufwändig experimentell zu untersuchen sind, können Computersimulationen hier zum besseren Verständnis ihrer Eigenschaften beitragen. Um die Elektronenstruktur von Molekülverbänden aus tausenden von Atomen wie Kohlenstoffnanoröhren zu beschreiben, kann man einfache quantenmechanische Methoden, gekoppelt mit Moleküldynamiksimulationen, anwenden. Damit lässt sich z.B. die Polarisation des Systems bei Ladungstransportphänomenen untersuchen und seine Leitfähigkeit berechnen.
Protein-Ligand-Wechselwirkungen und Medikamentenentwicklung
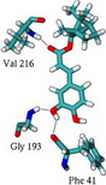 Ein zentrales Problem der Medikamentenentwicklung ist es, neue Substanzen zu finden, die biochemische Stoffwechselwege und Signalketten effizient und spezifisch beeinflussen. Da man nicht jede denkbare Substanz synthetisieren kann, ist es hilfreich, potentielle Liganden zunächst nur theoretisch zu untersuchen. In solchen „Virtual Screening“-Anwendungen geht es darum, die Bindungsgeometrien und -stärken neuer Substanzen an Zielproteine vorherzusagen. Im Idealfall lassen sich durch gezielte Veränderung der Molekülstruktur am Rechner verbesserte Inhibitoren designen die dann zu neuen Medikamenten weiterentwickelt werden können.
Ein zentrales Problem der Medikamentenentwicklung ist es, neue Substanzen zu finden, die biochemische Stoffwechselwege und Signalketten effizient und spezifisch beeinflussen. Da man nicht jede denkbare Substanz synthetisieren kann, ist es hilfreich, potentielle Liganden zunächst nur theoretisch zu untersuchen. In solchen „Virtual Screening“-Anwendungen geht es darum, die Bindungsgeometrien und -stärken neuer Substanzen an Zielproteine vorherzusagen. Im Idealfall lassen sich durch gezielte Veränderung der Molekülstruktur am Rechner verbesserte Inhibitoren designen die dann zu neuen Medikamenten weiterentwickelt werden können.
